Am. J. Hum. Genet. 64:118-125, 1999
Résumé :
Le syndrome de Williams (SW) est dû à une micro délétion d’approximativement 1.5 millions de bases dans une des deux paires du chromosome 7. Le tableau suivant fait le résumé des gènes supprimés dans cette région du chromosome :
|
Gènes
|
Protéine
produite par le gène |
Rôle
de la protéine |
|
ELN
|
Elastin
|
Il
s’agit d’une protéine très importante, qui intervient dans beaucoup d’organes, et dont le rôle est lié à l’élasticité des tissus. |
|
LIMK1
|
Tyrosin |
C’est
une protéine qui est exprimée dans le cerveau en développement. That phosphorylates and inactivates cofiline, a protein that is required for turnover of actin filaments. Actin depolymerization and recycling are required at the leading edge of a moving cellular process, so that defects in actin turnover could affect axonal guidance during CNS development. Since LIMK1 is expressed at high level in the CNS, it is a promising candidate for the mental aspects of the WS phenotype. |
|
STX1A
|
Syntaxin
1A |
Un
composant du dispositif synaptique (cerveau). |
|
RFC2
|
Code
pour une sous unité du complexe de réplication “facteur C” impliqué dans la réplication de l’ADN. |
|
|
FZD3
|
Homologous
to the Drosophila tissue-polarity gene “frizzled”. |
Cette micro délétion a donc fait disparaître plusieurs copies de certains gènes sur ce chromosome, la deuxième copie de ces gènes étant toujours disponible sur le second chromosome de la paire. Pour la plupart des gènes une réduction de 50 % de la production due aux gènes (haploinsuffisance) ne conduit à aucun effet visible (phénotype). Dans le cas du syndrome de Williams plusieurs anormalités physiques, cognitives et de comportement sont cependant souvent observées. Les phénotypes dus à des effets de dosage de gènes dépendent souvent des propriétés d’autres gènes non directement impliqués. Ceci conduit donc dans le cas du syndrome de Williams à une large variation de l’intensité des symptômes suivant les malades. Dans cet article une étude a été entreprise pour essayer de déterminer les effets (phénotype) de l’haploinsuffisance de chacun des gènes impliqués dans le cas du SW. Quatre patients ont été étudiés, ceux-ci ne présentent qu’une partie des symptômes de SW, le tableau suivant en fait le résumé.
|
Nom
des patients |
Symptômes
|
|
PM
|
Sténose
aortique supravalvulaire (SVAS) Hernie inguinale Classe d’éducation spéciale |
|
TM
|
SVAS
Glycosuria |
|
CS
(7 ans 8 mois) |
SVAS
Sténose artérielle pulmonaire Constipations fréquente pendant l’enfance |
|
HG
|
SVAS
|
|
Type
d’analyse de l’ADN |
Cibles
|
|
Analyse
de microsatellites (petites séquences répétitives d’ADN). |
7
Généthon primers + microsatellites de l’intron 18 de ELN + microsatellites de l’intron 13 de LIMK1 |
|
Méthode
FISH (using cosmid clones) |
ELN
LIMK1 STX1A |
|
Deletion
mapping of hybrids by PCR |
By
PCR, using primers designed to amplify the 5′ and 3′ regions of the genes ELN LIMK1 STX1A FZD3 RFC2 D7S489 |
|
Nom
du patient |
Analyse
Microsatellites |
Méthode
FISH |
Hybrids
par PCR |
|
PM
|
LIMK1
ELN |
LIMK1
|
pas
testé |
|
TM
|
LIMK1
ELN |
LIMK1
|
LIMK1
ELN à partir de l’exon 10 |
|
CS
|
beaucoup
voir figure 1 |
LIMK1
ELN STX1A |
LIMK1
FZD3 STX1A ELN RFC2 D7S489U(B) |
|
HG
|
beaucoup
voir figure 1 |
LIMK1
ELN |
LIMK1
ELN |
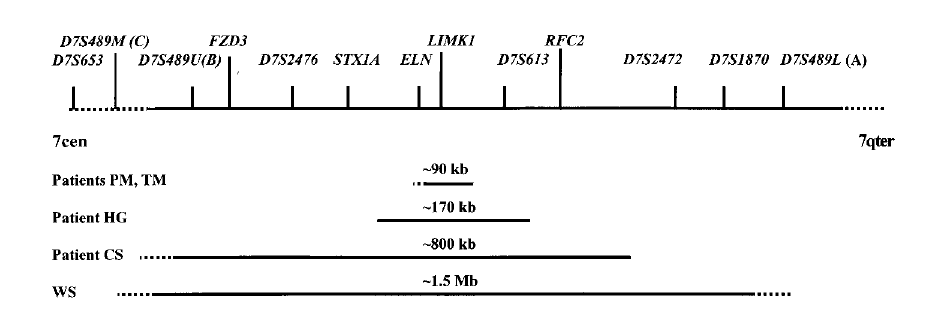
Figure 1 : Zone supprimée chez les différents patients. (extrait de l’article)
En plus des tests d’ADN, les patients ont subi des tests psychométriques (BAS II). Il s’agit d’une espèce d’évaluation de QI (intelligence). A partir de ces tests psychométriques ils peuvent tester 4 conditions (4 critères), c’est ce qu’ils nomment WSCP (WS cognitive profile), c’est plus adaptée à détecter le syndrome de Williams (en réalité indépendante du QI). Les personnes atteintes du SW sont très souvent positives pour ce test.
Aucun des patients étudiés n’a été détecté positif (WSCP) par ces tests.
Conclusion
Il est très intéressant de remarquer que ces patients, dont des parties plus ou moins grandes du chromosome 7 ont été supprimées, n’ont que peu de caractéristiques du syndrome de Williams (en particulier pas le visage, ni les caractéristiques intellectuelles). Seul le gène ELN est responsable des caractéristiques cardiologique, SVAS et hernie. Les gènes LIMK1 ou STX1A ne contribuent pas aux phénotypes du SW. Les autres symptômes caractéristiques du SW doivent être recherchés dans d’autres gènes que ceux étudiés dans cet article.
